本文是一篇计算机论文,本文针对实验数据进行了测试。模型设计和训练的最终目的是服务与实际应用,本文将其他文献中搜集到的真正经过实验验证的环形 RNA 数据作为测试数据集,使用本文效果最好的模型 DeepCircRNA 进行识别,准确率高达 83%。这个准确率说明,本实验所使用的模型可以真正的作为研究环形 RNA 的科研人员的有效工具。
1 前言
1.1 研究背景及意义
早在四十多年前 circRNA 就已经被发现报道,1976 年 Sanger H 等人就在植物类病毒中发现了环形的 RNA 分子(Sanger H.L et al.,1976);1979 年,Hsu 等人在电子显微镜下观察 HeL a 细胞,在 RNA 中有 1%-2%的数量为环形 RNA(Hsu et al.,1979);1991 年,Nigro 等人在肿瘤抑制基因中发现了异常剪接,表明外显子不总是按照顺序剪接,通过这种异常剪接创造出新的 RNA 产物(Nigro JM et al.,1991);1992 年,Cocquerelle 等人在人的 c-ets-1 基因中识别出一种新型的转录本,外显子的顺序是被打乱的,表达水平低于正常转录本(Cocquerelle C et al.,1992);1993 年,Capel B 等人在小鼠睾丸的 Sry 基因中发现了环形 RNA(Capel B et al.,1993)。至此,由于技术原因和环形 RNA 本身表达量较低,环形 RNA 被认为是异常可变剪接造成的 RNA 副产物,甚至也有人认为是由于实验过程中某些操作等人为原因引起的这种现象,所以一段时间以来环形 RNA 并没有引起相关领域的重视。
随着 RNA-seq 等生物信息技术的发展,人们获取了大量转录本数据的同时发现环形 RNA 并不像之前认为的是一种异常,意外的现象。Danan M 等人建立一种特异性,敏感的 RNA-seq 识别环形 RNA 的方法,在古细菌中发现了环形 RNA 普遍存在(Danan M et al.,2011);Salzman J 等人通过对正常和恶性人类细胞 RNA 深度测序,发现数百个基因的剪接转录本中很大一部分是环形 RNA,认为这是一种细胞中的普遍现象(Salzman J et al.,2012);Wilianm R jeck 等人利用高通量测序技术在人成纤维细胞中发现了超过 25000 个环形 RNA,并认为环形 RNA 可作为内源性竞争 RNA 发挥作用,是保守的,稳定的,可能参与控制基因表达(Jeck WR et al.,2013 )
环形 RNA 真正引起学术界的重视是因为两篇发表在 Nature 上的文章,Sabastian 等人对人,小鼠和线虫的 RNA 进行了测序分析,检测到了上千种表达良好并且稳定的环形 RNA,同时这些环形 RNA 具有组织和发育阶段的特异性表达,并且发现人的一个环形 RNA 被 microRNA 的效应物紧密结合,通过进一步的实验证明环形 RNA 形成大量转录后调控因子,具有以前未被认知的编码序列调节潜力(Sebastian et al.,2013);Hansen 等人发现环形 RNA 作为 microRNA 的海绵作用,CRIS-7 含有 70 多个选择性调控的 mirna 的靶位点,强烈抑制 miR-7 的活性,并且进一步证明了睾丸特异性表达的环形 RNA 是 miR-138 的海绵,这是史上第一次对自然的环形 RNA 做出了功能上的分析(Hansen et al.,2013)。
1.2 国内外研究现状
国内,白杨提出使用随机森林进行基因剪接位点的识别(白杨,2010);李绍燕提出概率统计模型对基因剪接位点预测(李绍燕等,2011);李琴等通过位置关联权重矩阵以及序列组分的多样性增量进行基因剪接位点的识别(李琴等,2014)。近年来,基因识别有了长足的进步,包括对传统方法的改进与新兴方法的应用。李素等使用群智能算法,改进 SVM 参数的选取方法(李素等,2018)。在人工智能技术中,深度学习是近年来成长最为迅速的,并且逐渐渗透到各个领域,其中基因识别领是其很重要的一个分支。YiLi 等人尝试用深度学习的方法做基因表达预测并取得了很好的效果,他们通过 1000 个 landmark 基因已知的基因表达值进行推算剩余的 21000 个 target基因,(Li Y et al.,2016;陈华宇,2017)。李洪顺等提出一种深度学习模型,可通过序列信息来预测 RNA 结合蛋白(李洪顺等,2018),邵丽芬则利用深度学习模型来解决蛋白质的分类问题(邵丽芬,2018)。
目前识别环形 RNA 的方法可以分为两大类,一种是基于 RNA-seq 数据的比对方法:2010 年,Wang 等人提出了一种基于 RNA-seq 的方法 Mapsplice 用于检测非规范拼接和新规范拼接,此方法不依赖于剪接位点特征和内含子长度(Wang K et al.,2010);2014 年,Zhang 等人设计了一种基于 RNA-seq 比对的方法 circexplorer 来发现环形 RNA 的剪接位点(Zhang XO et al.,2014);2015 年,Sazabo 等人设计了一种基于RNA-seq 的统计学方法 KNIFE 来检测环形 RNA,此方法不依靠 read 数和外显子同源性(Sazabo L et al.,2015);同年,Gao 等人提出了一种基于切屑剪裁信号的方法 CIRI 针对 RNA-seq 数据来对环形 RNA 进行不偏倚,准确的检测,首次识别并验证了内含子/基因间环形 RNA 特有片段在人类转录组中的流行程度(Gao Y et al.,2015);2016年 Tian 等人提出了一种可以对单端和双端 RNA 数据进行环形 RNA 丰度检测的方法 Acfs,具有识别融合环形 rna 的能力(You X T at al.,2016). 另一种是利用机器学习对RNA-seq 数据的序列特征进行学习预测:2015 年 Pan 提出了 PredcircRNA 一种基于机器学习的多核学习去区分环形 RNA 和其他 lncRNA,准确率为 0.778(Pan X Y et al.,2015);2018 年 Chen 提出了一种基于分层极限学习机 H-ELM(hierarchical extreme learning machine)的方法来区分环形 RNA 和其他 lncRNA,准确率为 0.789(Chen L et al.,2018);2019 年 Chaabane 等人提出了基于端到端的深度学习的方法,叫做 circDeep,也是区分环形 RNA 和其他 lncRNA,准确率达到 0.893(Chaabane L et al.,2019);2020 年,Niu 提出了一种将极限学习机与粒子群优化算法相结合的模型,名为 CirR NAPL,来区分环形 RNA 和其他 lncRNA,环形 RNA 和蛋白结合 RNA,干细胞中表达的环形 RNA 和非干细胞中表达的环形 RNA,准确率分别为 0.815,0.802,0.782(Niu M T et al.,2020)
2 基础知识
2.1 基因剪接位点基础概述
2.1.1 基因概述
基因是一段 DNA 片段,它是存储,传递遗传信息和复制细胞的主要物质。这些DNA 片段会转录成 RNA,然后 RNA 进一步翻译成各种蛋白质,来行使各种生物功能(杨福兰等,2014).
DNA 是由正负双链构成,每一条链都包含四种脱氧核糖核苷酸:腺嘌呤,胸腺嘧啶,胞嘧啶,鸟嘌呤,分别用字符‘A’,‘T’,‘G’,‘C’代表。它们不同的组合形成的序列也就代表着不同的 DNA 片段(郑大军,2014)。RNA 是由 DNA 的某一条链转录而来,所以是单链结构,由四种核糖核苷酸组成:腺嘌呤,尿嘧啶,胞嘧啶,鸟嘌呤,分别用‘A’,‘U’,‘G’,‘C’代表,RNA 中的‘A’由 DNA 中的‘T’转录而来,‘U’由‘A’转录而来,‘G’由‘C’转录而来,‘C’由‘G’转录而来。
2.1.2 基因结构
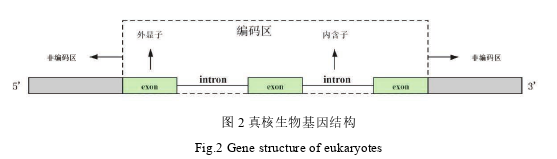
本文使用的数据均来自真核生物,则以真核生物的基因结构为例,如图 2 所示。其基因结构可以分类两大区域,编码区和非编码区,其中编码区包含外显子‘exon’和内含子‘intron’,外显子可以编码蛋白质,内含子不编码蛋白质。N 个外显子由 N-1 个内含子分隔开,一般情况下内含子的长度要远大于外显子。
2.2 非编码 RNA 简介
生物体中包含的 RNA 种类多,功能复杂,根据其是否编码蛋白质分为两类:编码RNA(coding RNA)和非编码 RNA(non-coding RNA)。前者指的是 mRNA,通过翻译的蛋白质行 使功 能,后 者包括:rR NA,tRNA,snRNA,snoRNA,piRNA,microRNA,circRNA 等,这些 RNA 都是从基因组转录而来,不需要翻译蛋白质,在RNA 水平上就可以发挥生物学功能。根据非编码 RNA 的长度一般可以分为三类:小于 50nt 的,如 microRNA,piRNA 称为 tiny no-coding RNA;介于 50nt 和 200nt 之间的,如 rR NA,tRNA 称为 small no-coding RNA;大于 200nt 的,如 circRNA 称为 longno-coding RNA(lncRNA)。
非编码 RNA 在生物体内发挥重要的生物功能。例如:rR NA 是生物体内含量最高的非编码 RNA,它和蛋白质结合形成核糖体,在 mRNA 的指导下将氨基酸合成为肽链,发挥蛋白质生物合成的“装配机”作用;snR NA 可以与蛋白因子结合形成小核糖蛋白颗粒,从而行使剪接 mRNA 的功能;microRNA 可以与 mRNA 结合,从而使 mRNA降解或者沉默。
近几年,随着人们对非编码 RNA 的研究深入,发现其与癌症有密切关系。非编码RNA CASC2 可以抑制直肠癌细胞的侵袭,迁移,增值,并且可能与靶基因 miR-514b-5p 有关,从而可提供治疗和预防的新的基因靶点(李豪等,2020);非编码 RNASNHG16 通过与靶基因 mi-16-5p 的作用影响胃癌细胞的凋亡和增值,可作为胃癌治疗和诊断的分子靶点(常利普,2020);非编码 RNA SNHG5 在缺氧诱导的情况下影响肝癌细胞的侵袭和迁移(李雅睿等,2020);乳腺癌的治疗和 lncRNA 的表达密切相关,Godinho 等发现,塔莫细分的耐药性和乳腺癌中的 BCAR4 通过靶向 ERBB2/HER2参与有关(Godinho MF et al.,2010)。
3 数据的获取与预处理····························· 15
3.1 原始数据的获取································· 15
3.1.1 正数据集的获取·····································15
3.1.2 负数据集的获取······························16
4 基于深度学习的环形 RNA 识别································· 20
4.1 模型设计·················· 20
4.1.1 基于 CNN 的剪接位点特征提取································· 21
4.1.1 基于 LSTM 的剪接位点特征提取·································· 22
5 环形 RNA 剪接位点在线识别工具的设计与实现 ···················47
5.1 系统设计 ···························47
5.2 识别模块 ··································48
5.3 比对模块 ····························48
5 环形 RNA 剪接位点在线识别工具的设计与实现
5.1 系统设计
本文的人环形 RNA 剪接位点在线识别系统前端采用 HTML,JavaScript 和 CSS,后端使用 python 语言。前端部署在阿里云服务器,后端部署在实验室的服务器上。系统设计图如图 42 所示。
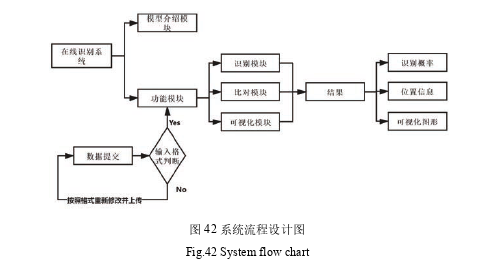
该系统共包含两大模块,其中模型介绍模块位于网页的正上方,对于该系统和模型做了简单的介绍,还介绍了此系统的目的和意义,让进入该网站的人对该系统有个大概的了解。用户操作模块是系统的核心模块,主要包含三大功能:识别功能,这是用户操作模块中的核心功能,对用户输入的单组数据或者上传的文件进行识别;比对功能,该功能可以帮助比对用户提交的序列,得到具体的位置信息;可视化功能,该功能可配合前两个功能利用可视化图形帮助用户更直观的理解。在数据提交到用户操作模块之前,对于用户输入的数据或者上传的文件会先进行检查,判断格式是否符合要求,模型接收的输入序列的长度为 80bp,若大于或者小于 80bp,均为非法输入,并且由于 DNA 序列理论上只会包含‘A’,‘T’,‘G’,‘C’在内的四个字符,若含有空格,数字等非 26 个大写英文字母出现时,同样也会判断为非法输入,会直接跳转到提示页面,要求检查输入格式,重新输入或者上传。
6 结论与展望
6.1 主要研究结论
随着实验的发展,环形 RNA 在各种生物体内大量发现,其生物功能和机制也被慢慢揭示。环形 RNA 作为 miRNA 的海绵,抑制 miRNA 与靶标结合,通过这种相互作用的机制来调节人体的各种生物功能;环形 RNA 还具有高保守性,稳定性的特点,可作为转录后调控因子。目前大量研究发现环形 RNA 与人类的重大疾病密切相关,尤其是癌症。如果可以揭示出环形 RNA 与癌症之间的秘密,那将推动整个人类健康事业的进步。本文对国内外环形 RNA 的相关研究进行了大量跟踪,在了解了环形 RNA 的相关生物原理后,提出了针对人和拟南芥的环形 RNA 剪接位点识别研究。现将本文的主要研究结论总结如下:
(1)数据的获取和针对性的预处理。虽然实验证明环形 RNA 在生物体内大量存在,但是公开发表的环形 RNA 数据和数据库并不多,特别是理想状态下的负数据集。对于人的环形 RNA 的数据是下载自目前使用最多,可信度最高的公开数据库CircBase,拟南芥环形 RNA 数据则是下载自世界研究研究植物环形 RNA 的三大数据库AtCircDB,PlantCircDB 和 PlantCircNet,然后进行比较,选择三者共有的数据作为正数据集。负数据集则是通过注释文件和数据库进行比较,筛选出的相对理想的数据。由于“ATGC”这种字符串形式的基因序列并不能直接作为深度学习模型的输入,针对不同物种的基因特性,选择适当的长度,从剪接位点这一角度出发,统一进行编码处理,构造成适合模型接受的输入形态。
(2)对比不同影响因子,选择出最优模型。本文选用卷积神经网络和循环神经网络中的 LSTM 作为模型中最重要的两部分,前者可以很好的学习编码后类似图片的二维结构的序列特征,后者可以结合序列前后部分的语义,更好的理解每一条序列,二者相结合可以更好的达到识别基因序列的目的。但是影响模型最终性能的因子还有很多,如卷积核大小,卷积深度,LSTM 数量,池化层的类型,序列之外的自变量,编码方式,序列长度,数据质量等。卷积核本文共尝试了 8 种大小,卷积深度测试了 2种,LSTM 数量测试了 3 种,共 16 个模型;又分别针对这 16 个模式分别采用均值池化和最大池化进行了测试;根据其他文献中提到的 GC 含量可能是影响成环的因素,从而加入了 GC 含量这一自变量,与序列共同进行训练;
参考文献(略)
