本文是一篇计算机论文,本文以构建基于网络表征的蛋白质相互作用预测模型为目的,针对仅依赖蛋白质序列信息的蛋白质相互作用预测模型的缺点,提出了将蛋白质序列信息和蛋白质相互作用网络结构相结合的预测模型。
第一章绪论
1.1研究背景及意义
人类基因组计划为生物学、医学等学科的发展做出了巨大的贡献,揭开了人体基因组及蛋白质序列的秘密,其中的测序工作为蛋白质的研究提供了宝贵财富,打开了通往蛋白质研究新领域的大门。蛋白质是人体的重要组成成分,几乎参与了生物生命活动的所有过程。然而大多数蛋白质都不是单独工作的,而是通过与其它蛋白质之间的相互作用(Protein-protein Interactions,PPIs)形成复合物进行协同工作。因此在探索蛋白质在生物体内的作用机制时,需要对PPI网络的开展深入的研究。蛋白质相互作用形成机制是现代分子生物学领域的重要研究课题之一。蛋白质相互作用可以调节生物体的大部分生命活动,包括新陈代谢、细胞间的通讯、基因转录[1]。为了能够直观地表示蛋白质间的相互作用关系,研究人员通过多个蛋白质之间的相互作用构建了PPI网络,从而更加深入地了解蛋白质工作原理,解析疾病等特殊生理状态下信号和能量物质代谢的机制,以及阐明不同蛋白质之间的功能联系,这些对于研究人员了解生物系统的运行有着十分重要的意义[2,3]。蛋白质之间的相互作用如果被改变甚至被破坏,则可能会引发细胞的功能障碍,造成严重后果。同时,针对PPI网络的研究和分析,对于新药研发人员寻找潜在的药物作用靶点也有着巨大帮助,为新药研发提供数据基础和理论依据。
随着科学技术的快速发展,研究人员提出了一系列用于检测蛋白质相互作用的技术,如质谱分析[4]、蛋白质芯片技术[5]、荧光共振能量转移[6](FRET)和酵母双杂交[7](Y2H)等。尽管这些生化实验方法促使了蛋白质相互作用数据成指数级别的增长,但蛋白质的功能注释及其结构数据更新速度较慢,而且通过生化实验方法获得的数据还存在误报、漏报、涵盖面窄等问题。现如今已知的大部分蛋白质相互作用关系都是通过生化检测实验得来的,但这些数据大约只占整体蛋白质相互作用数据的百分之二十左右。同时,研究人员在做检测实验时,会更加关注于那些功能较为重要的蛋白质同其它蛋白质的相互作用,这也就导致了蛋白质相互作用数据极具有偏向性。除此之外,研究人员还有可能会受到设备分辨率的限制以及操作过程中环境因素的影响,从而使得观察结果存在偏差导致假阳性和假阴性数据的产生。
1.2国内外研究现状
蛋白质是由一串氨基酸组成的大分子,在生物体内很少单独发挥作用,大部分情况下都是通过与其它蛋白质相互协调共同发挥作用,如果我们能够全面的认识整个PPI网络,这对蛋白质组学以及生物学来说都是极为重要的。研究人员针对蛋白质相互作用预测的研究已有几十年的历史,到目前为止,大致可分为两种方法,一种是生化实验方法,一种是计算方法。生化实验方法包含串联亲和纯化(TAP)[12]、下拉测定法(Pull-down)[13]、免疫共沉淀[14]、和生物层干涉仪(BLI)[15]等,这些方法为蛋白质组学的发展做出了极大的贡献,使得人们对PPI网络有了更清晰的理解。但是这些方法需要通过大量的实验来反复验证蛋白质之间相互作用关系,极为耗时耗力。同时,由于技术的局限性导致这些方法或多或少存在一些缺陷,如实验设计不合理、无法完全模拟蛋白质在人体内活动的环境以及实验过程中存在的数据误差等,较易产生大量的假阳性和假阴性数据。使用生化实验方法对蛋白质相互作用进行验证不仅要花费大量的人力、物力、财力,而且验证的效率还不高,到目前为止通过生化实验验证的蛋白质相互作用仍然是整个蛋白质相互作用数据集的冰山一角[16]。因此对于PPI预测的研究开始转向计算方法,并逐渐成为了研究蛋白质相互作用的主流方法[17-20]。目前较为流行的预测蛋白质相互作用的方法大致可以分为四类,包括基于蛋白质一级结构的方法、基于蛋白质三级结构的方法、基于PPI网络的方法和基于带属性信息的PPI网络的方法。
第二章相关知识
2.1蛋白质相关数据库介绍
(1)DIP数据库
数据库中的蛋白质相互作用数据都是经过生化实验验证过的。该数据库结合了来自各种来源的信息,创建了一组单一的蛋白质相互作用数据集。存储在DIP数据库中的数据不仅由专业的管理人员手动管理,同时还使用计算方法进行自动管理,这些计算方法从DIP数据核心子集中,提取有关蛋白质相互作用网络的知识,从而保证数据的可靠性。
(2)BioGRID数据库
BioGRID是一个生物医学交互存储库,用于保存和分享各种生物的遗传和蛋白质相互作用的数据,目前BioGRID数据库拥有1740000条蛋白质相互作用数据,这些数据要么是经过高通量技术检测得来的,要么就是经过研究发现的。数据库记录了包括酿酒酵母(Saccharomyces cerevisiae)、裂殖酵母(Schizosaccharomyces pombe)和拟南芥(Arabidopsis thaliana)等其他物种的蛋白质相互作用数据,BioGRID数据库的搜索功能也极为强大,可以跨数据类型和来源进行快速查询,并且所有数据集都是免费向用户提供的,并且可以使用多种标准化格式下载。所有BioGRID的文档都可以在BioGRIDWiki中在线获得。
(3)MINT
MINT数据库中的数据主要依赖于专家从科学文献中的挖掘,并且挖掘出来的都是经过实验验证的数据。数据库记录大肠杆菌、幽门螺杆菌等六百多个物种的蛋白质相互作用。针对该数据库相关的出版物有6000多个,一共有130000多条蛋白质相互作用数据。MINT数据库与26000多位研究人员有过信息交互。
2.2蛋白质序列的编码方式
(1)氨基酸组成编码
氨基酸组成编码(AAC)是一种较为基础的编码方式,通过计算蛋白质序列中20种天然氨基酸(即ACDEFGHIKLMNPQRSTVWY)的频率对蛋白质进行编码。
(2)联合三元组编码
联合三元组编码(CT)提出将任意三个连续的氨基酸视为一个单元来设计蛋白质序列编码的方式,同样是通过计算频率对蛋白质进行编码,不同的是联合三元组编码是计算三个连续氨基酸在序列中出现的频率。首先将20种天然氨基酸根据其侧链基团和偶极子分为七类,然后三个连续的氨基酸会有7*7*7=343种不同的类型,固定一个大小为3的窗口从序列的头划到尾,统计这343种类型出现的频率。原理上,如果蛋白质序列越长频率就会越大,因此还需要进行归一化操作。
(3)Quasi–Sequence–Order编码
Quasi–Sequence–Order(QSOrder)编码方式与前面介绍的两种方法有很大的不同,QSOrder编码是一种考虑氨基酸种类和氨基酸排列顺序的编码方法,不仅如此该编码方式还利用了氨基酸的物理性质(包括亲水性,疏水性,侧链基团等)和化学性质[43]。QSOrder编码方法通过一组序列耦合数来从蛋白质序列中提取特征信息。
第三章基于图神经网络的PPI预测模型....................................17
3.1基于变分图自编码器PPI预测方法..........................17
3.1.1算法流程.............................17
3.1.2数据准备......................18
第四章基于拓扑特征和生物信息的PPI预测模型............................26
4.1基于拓扑特征和生物信息的PPI预测方法..............................26
4.1.1网络拓扑结构分析..............................................27
4.1.2蛋白质相互作用规则............................................27
第五章总结与展望............................35
5.1总结..............................35
5.2展望................................35
第四章基于拓扑特征和生物信息的PPI预测模型
4.1基于拓扑特征和生物信息的PPI预测方法
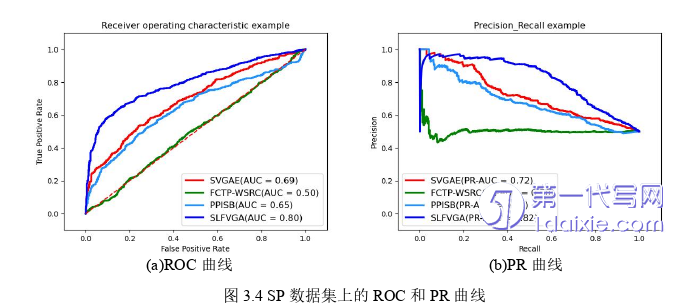
蛋白质相互作用网络在蛋白质组学研究中起着至关重要的作用。然而,可用的蛋白质相互作用数据仍然不够充足。正是出于这个原因,研究者们提出了各种算法来预测蛋白质相互作用网络,大部分都是基于蛋白质序列的预测方法。尽管这些方法有效,但它们的性能会受到蛋白质相互作用数据中假阳性和假阴性数据的限制。为了克服这个问题,本章提出了一种新的蛋白质相互作用网络预测算法即PASNVGA,该模型首先采用不同的策略从蛋白质的序列信息和网络结构中提取特征,设计了一个评分函数来衡量蛋白质之间的高阶连通性,从而得到一个高阶邻接矩阵,接着利用结合了蛋白质序列信息和高阶邻接矩阵的变分图自编码器对蛋白质相互作用网络进行网络表征。同时我们还分析了多种传统分类器对预测结果的影响,经过比较最后我们选择了较为简单的前馈神经网络作为蛋白质相互作用网络预测模型的分类器。为了选择更契合模型的蛋白质编码方式,我们将传统的蛋白质编码方式和QSOrder编码方式进行对比,发现使用QSOrder编码方式预测模型有更好的性能。我们做了大量的实验并将其与几种先进的模型作对比,实验结果表明,我们的模型在预测蛋白质相互作用上有着十分出色的性能。
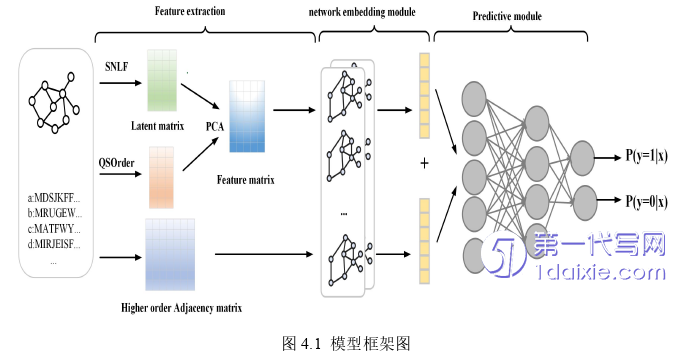
本章构建的改进后的预测模型框架如图4.1所示。
第五章总结与展望
5.1总结
蛋白质相互作用网络也是近些年来研究的热点,研究蛋白质相互作用网络对药物研发和疾病诊断都有极大的推动作用。使用生化实验方法检测蛋白质相互作用还存在很大的缺陷,因此使用计算方法预测蛋白质相互作用网络是最可靠的选择,另外现在的蛋白质相互作用网络还是一个高维稀疏的网络,使用传统机器学习方法很难满足预测的需求。本文以构建基于网络表征的蛋白质相互作用预测模型为目的,针对仅依赖蛋白质序列信息的蛋白质相互作用预测模型的缺点,提出了将蛋白质序列信息和蛋白质相互作用网络结构相结合的预测模型。模型整体上可以分为两个部分,第一部分是特征提取,通过图神经网络将蛋白质相互作用网络中的蛋白质节点表征成低维特征向量。第二部分结合蛋白质表征向量选择合适的分类器预测蛋白质相互作用。本文提出了两种基于网络表征的蛋白质相互作用预测方法,第二种方法可以说是在第一种方法的基础上做了改进,并且取得了很好的效果。论文的主要内容包括以下几个方面:
(1)构建了基于蛋白质序列和属性信息的蛋白质相互作用预测模型,提出了使用对称非负隐特征模型从蛋白质相互作用网络中提取节点隐特征,减轻噪声数据带来的负面影响。为了能够获得更有效的蛋白质节点特征信息,将蛋白质节点特征和蛋白质序列特征通过主成分分析法有效的整合在一起。利用变分图自编码从网络结构信息和节点特征信息中进行网络表征。接着在网络表征的基础上,我们采用神经网络分类器来预测蛋白质相互作用,最终的实验结果也表明了本文提出的方法可以准确的预测蛋白质相互作用。
(2)设计了一种结合蛋白质序列信息、低阶邻居信息和高阶邻居信息的高阶邻接矩阵计算方法,该方法不仅结合了蛋白质相互作用理论,同时还使用了随机游走的思想。该计算方法一方面考虑了已知的蛋白质相互作用的影响,另一方面还考虑了潜在的蛋白质相互作用的影响。实验结果表明,结合了该计算方法的模型在预测性能上有着极大的提高,同时也为蛋白质相互作用预测方面的研究提供了新的方向。
参考文献(略)
