本文是一篇毕业论文范文,本论文的主要成果如下: (1)针对转录组原始数据,首先对其进行了质量控制,清除样本中测序质量较低的读段和碱基数量异常的读段。其次,根据玉米的参考基因组信息,对转录组测序数据中各个基因的表达水平进行了定量操作。之后,为了保证后续基因表达水平在组间比较时结果的合理性和准确性,研究了基因表达数据的标准化方法,并对基因表达计数信息完成了标准化操作。此外,对于表型数据中 14%水分标准产量与转录组测序数据不同期的情况,通过最佳线性无偏预测对测序样本表型信息的真实值进行了估计。
第1章 绪论
1.1 课题背景及研究的目的和意义
玉米最初种植于墨西哥,是世界各地最广泛种植的粮食作物之一,是世界上三成人口的主要食物,是人类营养、动物饲料和生物能源的重要来源。同时,玉米还具有极高的饲用价值和工业价值。因此,确保并提高玉米产量和质量显得极其重要。考虑到全球耕地面积的减少,提高玉米产量是确保粮食安全的重要举措[1]。此外,玉米作为一种模式作物,针对玉米进行生物信息学分析,能够促进对于基因组以及植物遗传学的理解,有助于分子育种。
玉米籽粒大小会影响籽粒灌浆、幼苗活力和最终使用质量,从而影响市场等级和消费者偏好。玉米籽粒所表现出的性状作为数量性状,其遗传机制复杂,受数量性状基因座(Quantitative Trait Locus,QTL)控制,且受环境变化的影响[2]。对玉米籽粒相关性状的遗传机制以及遗传基础进行探究,揭示玉米产量的优势等位基因,将有助于阐明玉米种子发育的调控机制,有助于制定提高玉米产量的策略,对玉米产量的遗传调控具有重要意义。
另一方面,随着高通量测序技术不断完善,玉米相关的各方面数据不断积累,尤其是针对转录组数据的研究越来越多。组织或细胞在特定发育阶段或功能状态下转录得到的全体 RNA 的总集被称为转录组。转录组分析是一个强大的工具,用于更好地了解控制宿主细胞命运、发展和疾病进展的潜在途径[3],同时,它还是一种迅速、高效的基因组调查、大规模功能基因和分子标记鉴定的方法[4]。转录组分析作为从整体层面研究基因功能和结构的分析方法,能够揭露特定生物学进程与疾病发展进程内的分子机理,为开展基因功能及结构研究奠定了基础。研究转录组是诠释基因组功能元件和揭露细胞及组织内分子组成的重中之重,同时对探究机体发育和疾病进展产生重大影响。
1.2 国内外研究现状
国内外针对玉米的生物信息学相关研究层出不穷。在产量性状方面,Raihan 等人[6]、MIN L 等人[7]分别对影响玉米产量的籽粒长度(KL)、粒宽(KW)和粒厚(KT)的遗传基础进行了研究,Raihan 等人[6]还对玉米百粒重性状进行了研究,检测到与其相关相关的 19 个数量性状位基因座,Y. Liu 等人[8]、Liu 等人[9]分别对产量相关的 QTL 和候选基因进行了检测;在玉米穗相关性状方面,张焕欣等人[10]、刘坤[11]、韩善冬[12]也分别进行了研究并得到了一些成果。此外,马兰等人[13] 、杨峥等人[14]、闫丽斌等人[15]、王玉莹等人[16]对玉米抗病性相关基因进行了研究,Guo 等人[17]对玉米水分胁迫下的转录组进行了分析。
针对玉米大量的生信研究,与高通量测序技术以及基因组、转录组分析方法的发展离不开关系。20 世纪 70 年代发表的 Sanger 法[18]奠定了人类全基因组计划的基础,由此开启了人们对于基因组的研究。之后,高通量测序技术的进步催生了基因芯片和基于RNA-Seq 的转录组测序等技术,并产生了大量的测序数据。转录组分析是一种迅速且高效的基因组调查、大规模功能基因挖掘和分子标记鉴定的方法[19]。相较于基因芯片等方法,基于 RNA-seq 的方法不依赖基因的先验知识,能够覆盖更大的转录组范围,具有更高的分辨率并且测序成本更低[20],已有很多学者针对 RNA-Seq 测序数据进行了研究[21, 22]。目前,常见的转录组分析方法主要可以分为两类:
1.2.1 基于统计学的假设检验
根据已知的生物学领域知识和统计学知识对数据进行处理,筛选出相对低维的RNA-Seq 数据进行后续研究,比较典型的方法是差异表达分析[23, 24]。差异表达分析是确定不同实验条件下基因表达的读段数,然后通过统计学检验比较具有显著表达差异的基因的方法。由于在生物体的各个组织和不同的发育周期内,各个基因的表达模式存在差异,因此可以通过检测样本中差异表达的基因减小候选基因的范围,初步确定感兴趣的基因。基于统计学的假设检验进行差异表达分析,通常使用 Fisher 精确检验[25]、似然比检验(Likelihood Ratio Test,LRT)[26]等。
第2章 转录组测序数据及表型数据预处理
2.1 转录组测序数据
2.1.1 高通量测序技术概述
高通量测序技术,是目前最新一代的基因测序技术,亦称做下一代测序(Next-generation Sequencing,NGS)技术。高通量测序技术的诞生,极大程度的降低了测序的成本,并且大幅提升了测序量,单次测序量能够达到几十万至上百万条 DNA 序列。此外,由于高通量测序技术具有极高的测序深度,因而也被叫做深度测序(Deep Sequencing)技术。高通量测序技术的诞生,催生了基因芯片和基于 RNA-Seq 的转录组测序等技术。特别的,RNA-Seq 技术能够测量低丰度转录物表达水平、测序覆盖范围大、能揭示转录物连接的精确位置和外显子连接点的优势,为相关学者进行基因表达、分子育种等生物信息学研究提供了大量高质量数据。
当下常用的高通量测序平台主要有 Roche 454 焦磷酸测序平台、Illumina/Solexa 合成测序平台和 ABI SOLiD 连接法测序平台等,其中以 Illumina/Solexa 平台使用最多。使用 Illumina/Solexa 进行测序需要经过制备 cD NA 文库、桥式 PCR 扩增、制备 DNA 单链和荧光信号分析等过程。
2.1.2 fastq 格式数据详解
通过 Illumina/Solexa 测序平台得到的原始文件(Raw Data)一般保存为 fastq 格式,每个 fastq 文件中包含若干个基因读段,每个读段分为四个部分,共有 4 行,具体如下图 2-1。

2.2 转录组测序数据预处理
2.2.1 质量控制
在 RNA-Seq 原始数据中,存在测序质量较低的读段以及测序长度异常的读段,比如测序长度小于 75 的读段;此外,读段中还存在衔接子(adaptor)序列,这些序列会影响之后的基因表达定量信息,需要通过质量控制的手段提前进筛选和清除。已经开发有许多用于进行 Raw Data 质量控制的工具,如 Fastqc、RSeQC 等。
2.2.2 构建索引
玉米作为一种模式作物,其参考基因组与参考基因组注释文件已经被制作完成,在Ensembl 网站(ftp://ftp.ensemblgenomes.org/pub/plants/release-44)能够获得上述文件的下载路径。由于本文数据集所测的玉米样本参考基因组还不确定,因此选用 B73 系玉米的参考基因组作为代替。需要指出的是,所用参考基因组信息对应的玉米品系不会对最终结果产生影响。
得到玉米参考基因组注释文件后,需要从中提取外显子和剪切位点。之后,根据所得外显子和剪切位点信息,使用参考基因组构建索引文件。该索引文件的作用是作为后续测序读段中碱基序列的对照模板,使得能够确定 RNA 序列的来源,即由哪一个基因进行转录而来。
第 3 章 结合 LASSO 的玉米 14%水分标准产量相关基因挖掘 .............. 18
3.1 最小绝对收缩和选择算法 ............................................ 18
3.1.1 最小绝对收缩和选择算法原理 ........................................ 18
3.1.2 最小绝对收缩和选择算法的解算法 ...................................... 19
第 4 章 融合遗传算法与 XGBoost 的玉米百粒重相关基因挖掘 ........... 34
4.1 遗传算法原理 .................................... 34
4.2 极限梯度提升算法原理 ................................ 35
4.3 基于遗传算法-XGBoost 的特征选择 .................................... 36
结论 ................................ 55
第4章 融合遗传算法与 XGBoost 的玉米百粒重相关基因挖掘
4.1 遗传算法原理
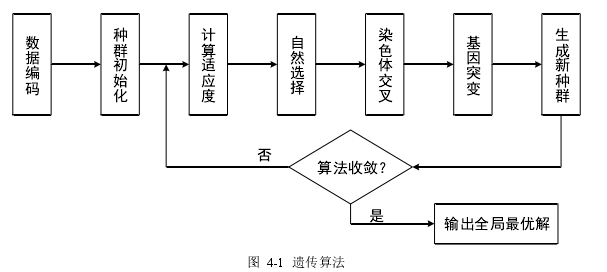
遗传算法(Genetic Algorithm,GA)借鉴了自然界中的物种进化的规律,是一种模拟达尔文生物进化论从而寻找最优解的随机搜索算法,最早由 J. Holland 发表在其专著《自然界和人工系统的适应性》[55]中。遗传算法每次迭代保留一组候选解,通过模仿生物繁殖的过程产生新的候选解集。
遗传算法是一个在全局层面对问题寻找最优解的算法。使用遗传算法搜索最优特征子空间的优势在于它不需要事先考虑相关的领域知识,并且由于每次迭代都是针对一个种群进行整体评价,因此能够考虑特征间的相互作用。
遗传算法的核心思想是优胜劣汰。通过编码将问题的解表示为遗传空间的个体,随机选取一定量个体作为最初的种群,通过不断的对种群进行演化迭代,每次迭代评估种群中所有个体对环境的适应性,保留适应度更高的若干种群并允许它们产生新一代种群,最终达到全局最优解。算法的实现流程如图 4-1 所示。
结论
玉米作为一种重要的农作物,保证玉米种植的高质高量是一项重要的研究课题。高通量测序技术的发展催生的基因芯片和转录组测序等技术,尤其是转录组测序技术的出现,加速了学者们从生物信息学角度研究玉米的进度。目前,针对玉米进行转录组分析,研究人员一般通过寻找组间差异表达基因,然后构建共表达网络、进行本体注释等方法锁定感兴趣的基因,这种方法依赖于已有的先验知识。此外,转录组样本测序数据具有极高的数据维度,这对数据分析方法提出了要求,如何降低数据处理阶段样本信息高维的影响、减少计算资源的消耗成为了一个重要的研究方向。
本文将挖掘影响玉米产量性状的基因作为切入点,从两组玉米转录组测序数据出发,探讨了针对 RNA-Seq 数据的转录组分析方法,提出了结合机器学习算法的方法,并用所提方法对玉米 14%水分标准产量和百粒重两个数量性状关联基因进行了研究,得到了36 个与玉米 14%水分标准产量性状相关的候选基因以及 48 个与百粒重性状相关的候选基因。论文的主要成果如下:
(1)针对转录组原始数据,首先对其进行了质量控制,清除样本中测序质量较低的读段和碱基数量异常的读段。其次,根据玉米的参考基因组信息,对转录组测序数据中各个基因的表达水平进行了定量操作。之后,为了保证后续基因表达水平在组间比较时结果的合理性和准确性,研究了基因表达数据的标准化方法,并对基因表达计数信息完成了标准化操作。此外,对于表型数据中 14%水分标准产量与转录组测序数据不同期的情况,通过最佳线性无偏预测对测序样本表型信息的真实值进行了估计。
(2)提出结合 LASSO 算法的转录组分析方法,以一组 389 个玉米自交系转录组数据作为实验数据集,研究了影响玉米 14%水分标准产量的基因。首先通过差异表达分析和皮尔森相关性分析从样本测序数据包含的全体基因中进行了初步筛选,得到由 2030个基因构成的基因子集。之后,使用该基因子集和转录组样本的最佳线性无线预测结果训练 LASSO 模型。利用 LASSO 算法能够完成数据降维的特点,在完成模型参数调优之后,最后从基因子集中筛选出 36 个与玉米 14%水分标准产量性状相关的候选基因,并对其进行了基因本体注释和 KEGG 通路分析。
参考文献(略)
